
翻訳後修飾によるシグナル伝達制御とその破綻に起因する疾患の数理モデル
 |
研究代表者 市川一寿 東京大学医科学研究所腫瘍数理分野 http://www.ims.u-tokyo.ac.jp/mathcancer/ |
【研究概要】
修飾シグナル病では異分野連携による研究を推進しています。これは異分野連携によって単独分野では明らかにできなかったことを明らかにしたり、異なる手法によって裏付けを補強したり、異分野間の議論によって新たな方向性を見出したりできるからです。私たちの計画研究では細胞内シグナル伝達の数理シミュレーションを介して実験研究と連携を推進しています。
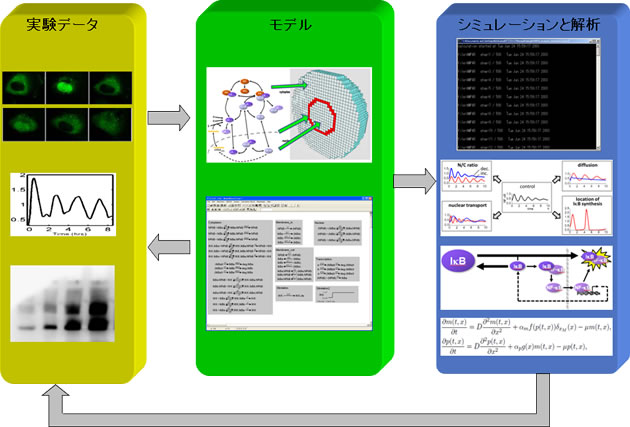
一例としてNF-κBのシグナル伝達についての異分野連携を紹介します。NF-κBはサイトカインなどの細胞外からの刺激によって活性化されて核移行し、プロモーター領域に結合して遺伝子発現に関与することはご存じのとおりですが、面白いことに核内NF-κBが周期2時間程度で振動することが知られています。しかも振動パターンの違いが遺伝子発現の違いを生むと考えられています。これは多くの数理・シミュレーション研究者の興味を引き、これまでに40以上の論文が出されています。ところがこれらは細胞の空間的広がりを考えておらず、細胞質と核の空間的分離すら考えずにすべてのシグナル伝達が1点で進行する「点モデル」でした。これは奇妙かつ不十分であることは言うまでもありません。そこで私たちは3次元的な広がりを持つ球形細胞をコンピュータに再現し、その中心に核-細胞質体積比(N/C比)を8.3%に設定した核を配置し、細胞質・核膜・核内にそれぞれ対応する反応式を割り付けてNF-κB振動のシミュレーションを行いました。こうすることでこれまで研究の対象にしてこなかったシグナル伝達の空間的パラメータの影響を知ることができるはずです。結果は私たちの予想を超えており、N/C比・核膜輸送(たとえば核膜孔の数によって変化します)・タンパク質の拡散定数(オルガネラの分布や密度で変化します)・タンパク質の翻訳場所の4つの空間パラメータすべてがNF-κBの振動パターンに影響しました。しかもその影響は予想をはるかに超えた大きなもので、空間パラメータが振動パターンの重要な制御因子であることを初めて示しました(Ohshima, PLoS ONE, 2012, e46911)。これは領域代表の井上先生との連携研究で、パスウェイ構築やデータ解析について連携しながら研究を進めています。
数理・シミュレーションは取つき難いと感じる実験研究者は多いと思います。私たちは誰でも使える(と信じています)シミュレーションソフトウエアA-Cellを公開しています (URLはこちら)。さらに主に若手研究者を対象に講習会を行ったり、数理シミュレーションの解説を領域HPに掲載したりしています(URLはこちら)。ぜひ一度ご覧下さい。もちろん異なる価値観や言葉が異分野連携の障壁となりますが、これは乗り越えられます。障壁の先にある新たな研究の展開を、一緒に目指したいと思います。数理・シミュレーションが使えるかもしれない、あるいは使いたいと思う実験の方々はお気軽に声をおかけください。
【参考文献】
- Watanabe, A., Hosino D., Koshikawa, N., Seiki, M., Suzuki, T., and Ichikawa, K., “Critical role of transient activity of MT1-MMP for ECM degradation in invadopodia”, PLoS Comp.Biol., Accepted.
- Ohshima, D., Inoue, J., Ichikawa, K., “Roles of Spatial Parameters on the Oscillation of Nuclear NF-κB: Computer Simulations of a 3D Spherical Cell”, PLoS ONE, 7 (2012), e46911.
- Hoshino, D., Koshikawa, N., Suzuki, T., Quaranta, V., Weaver, A.M., Seiki, M., Ichikawa, K., “Establishment and validation of computational model for MT1-MMP dependent ECM degradation and intervention strategies”, PLoS Comp.Biol., 8 (2012), e1002479.
- Saitou, T., Rouzimaimaiti M., Koshikawa, N., Seiki, M., Ichikawa, K., Suzuki, T., “Mathematical modeling of invadopodia formation”, J.Theor.Biol., 298 (2012), 138-146.
- Ichikawa, K., “Localized activation of proteins in a free intracellular space: dependence of cellular morphologies and reaction schemes”, BioSys., Vol.105(2011), 173-180.
- Ichikawa, K., Suzuki, T., and Murata, T., “Stochastic simulation of biological reactions, and its applications for studying actin polymerization”, Physical Biol., Vol.7(2010), 046010.
- Ichikawa, K., Hoshino, A. and Kato, K., “Induction of Synaptic Depression by High Frequency Stimulation in Area CA1 of the Rat Hippocampus: Modeling and Experimental Studies”, Neurocomputing, Vol.70(2007), pp.2055-2059.
- Imai, H., Kefalov, V., Sakurai, K., Chisaka, O., Ueda, Y., Onishi, A., Morizumi, T., Fu, Y., Ichikawa, K., Nakatani, K., Honda, Y., Chen, J., Yau, K.-W., and Shichida, Y., “Molecular Properties of Rhodopsin and Rod Function”, J.Biol.Chem., Vol.282(2007), pp.6677-6684.
- Ichikawa, K., “A Modeling Environment with Three-Dimensional Morphology, A-Cell-3D, and Ca2+ Dynamics in a Spine”, Neuroinformatics, Vol.3(2005), pp.49-64.
- Ichikawa, K., “A-Cell: graphical user interface for the construction of biochemical reaction models”, Bioinformatics Vol.17(2001), pp.483-484.











