
翻訳後修飾によるNF-κB活性化シグナルの制御機構と疾患発症との関連
 |
研究分担者 尾山 大明 東京大学医科学研究所 疾患プロテオミクスラボラトリー http://www.ims.u-tokyo.ac.jp/mpl/top.html |
【研究概要】
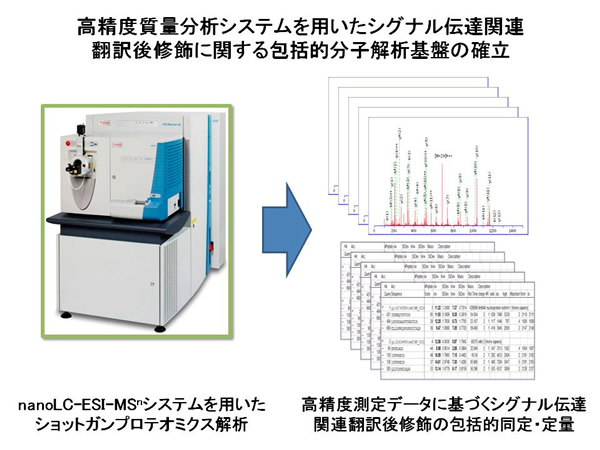
ゲノム科学に続いて次世代の生命科学研究における中核の一つとして注目されてきたプロテオミクス研究は、測定機器が発展途上であったこともあり、その進展は比較的緩やかなものであった。しかしながら高感度かつ包括的な解析を可能とするオンラインナノ流速液体クロマトグラフィーータンデム質量分析(nanoLC-MS/MS)システムの登場により、現在では数千のタンパク質の同定・定量を通してシステムレベルでのタンパク質の動態を俯瞰することが技術的に可能となってきている。東京大学医科学研究所附属疾患プロテオミクスラボラトリーにおいては、高精度のショットガンプロテオーム解析を可能とする次世代型nanoLC-FT-MSnシステムをはじめとして計6台の質量分析システムが設置され、従来行われてきたタンパク質の一次構造解析に止まらず、リン酸化、ユビキチン化、SUMO化、グリコシル化、アセチル化、メチル化などのあらゆる翻訳後修飾に関する包括的な構造決定を可能とする解析基盤を現有している。
また、近年の定量プロテオミクス技術の進展によって、シグナル伝達ネットワークをはじめとする細胞制御システムに関する時系列動態情報を包括的に計測することも不可能ではなくなってきており、当研究グループにおいてもSILAC(Stable Isotope Labeling by Amino acids in Cell culture)法に基づくin vivoタンパク質ラべリング技術と微量リン酸化シグナル因子群の特異的精製法を導入・開発することにより、細胞外部からの刺激の入力によって作動するリン酸化ネットワークに関する精密な活性動態計測技術を確立している。本研究計画においては、上記の先端プロテオミクス技術を用いて、NF-κB活性化シグナルをはじめとする多様な細胞内情報伝達経路に関与するシグナル複合体の既知あるいは新規の翻訳後修飾ダイナミクスを包括的に計測し、今まで各研究者によって分散的に行われてきた疾患シグナル研究に対して、システムレベルでの制御機構の解明に向けた統合的な解析プラットフォームの構築に貢献する。
本領域において、これまでにシグナル複合体構成因子あるいはその翻訳後修飾に関する高精度プロテオーム解析を推進し、井上純一郎先生(計画班)や深田吉孝先生(公募班)との異分野連携において共同研究成果の発表を行っている(Shibata et al., Nat. Commun., 2012; Hirano et al., Cell, 2013)。
【参考文献】
- Hirano A, Yumimoto K, Tsunematsu R, Matsumoto M, Oyama M, Kozuka-Hata H, Nakagawa T, Lanjakornsiripan D, Nakayama KI, and Fukada Y.
FBXL21 regulates oscillation of the circadian clock through ubiquitination and stabilization of cryptochromes.
Cell, 152: 1106-1118 (2013). - Shibata Y, Oyama M, Kozuka-Hata H, Han X, Tanaka Y, Gohda J, and Inoue J.
p47 negatively regulates IKK activation by inducing the lysosomal degradation of polyubiquitinated NEMO.
Nat. Commun., 3: 1061 (2012). - Kozuka-Hata H, Nasu-Nishimura Y, Koyama-Nasu R, Ao-Kondo H, Tsumoto K, Akiyama T, and Oyama M.
Phosphoproteome of human glioblastoma initiating cells reveals novel signaling regulators encoded by the transcriptome.
PLoS One, 7: e43398 (2012). - Oyama M, Nagashima T, Suzuki T, Kozuka-Hata H, Yumoto N, Shiraishi Y, Ikeda K, Kuroki Y, Gotoh N, Ishida T, Inoue S, Kitano H, and Okada-Hatakeyama M.
Integrated quantitative analysis of the phosphoproteome and transcriptome in tamoxifen-resistant breast cancer.
J. Biol. Chem., 286: 818-829 (2011). - Arii J, Goto H, Suenaga T, Oyama M, Kozuka-Hata H, Imai T, Minowa A, Akashi H, Arase H, Kawaoka Y, and Kawaguchi Y.
Non-muscle myosin IIA is a functional entry receptor for herpes simplex virus 1.
Nature, 467: 859-862 (2010). - Tasaki S, Nagasaki M, Kozuka-Hata H, Semba K, Gotoh N, Hattori S, Inoue J, Yamamoto T, Miyano S, Sugano S, and Oyama M.
Phosphoproteomics-based modeling defines the regulatory mechanism underlying aberrant EGFR signaling.
PLoS One, 5: e13926 (2010). - Fukui R, Saitoh S, Matsumoto F, Kozuka-Hata H, Oyama M, Tabeta K, Beutler B, and Miyake K.
Unc93B1 biases Toll-like receptor responses to nucleic acid in dendritic cells toward DNA- but against RNA-sensing.
J. Exp. Med., 206: 1339-1350 (2009). - Oyama M, Kozuka-Hata H, Tasaki S, Semba K, Hattori S, Sugano S, Inoue J, and Yamamoto T.
Temporal perturbation of tyrosine-phosphoproteome dynamics reveals the system-wide regulatory networks.
Mol. Cell. Proteomics, 8: 226-231 (2009). - Oyama M, Kozuka-Hata H, Suzuki Y, Semba K, Yamamoto T, and Sugano S.
Diversity of translation start sites may define increased complexity of the human short ORFeome.
Mol. Cell. Proteomics, 6: 1000-1006 (2007). - Oyama M, Itagaki C, Hata H, Suzuki Y, Izumi T, Natsume T, Isobe T, and Sugano S.
Analysis of small human proteins reveals the translation of upstream open reading frames of mRNAs.
Genome Res., 14: 2048-2052 (2004).











