
トップダウンリン酸化プロテオミクスを指向したアフィニティー磁気ビーズの創出
 |
研究代表者 木下英司 広島大学大学院医歯薬保健学研究院医薬分子機能科学研究室 http://home.hiroshima-u.ac.jp/tkoike/ |
【研究概要】
タンパク質のリン酸化修飾は,細胞内において,時間的・空間的に変化する極めて動的な現象であり,生命維持にかかわる重要な調節機構を担っています。よって,タンパク質のリン酸化部位を同定し,その動態を調べることは,リン酸化によるタンパク質の機能調節を明らかにする上で非常に重要です。最近では,生体内タンパク質を酵素消化した後のペプチド混合物からリン酸化ペプチドのみを濃縮する方法の開発,改良が進み,それと同時に質量分析装置のさらなる高度化が進んだ結果,タンパク質リン酸化部位の同定数は飛躍的に増大しました。また,濃縮したリン酸化ペプチドの量的な変化を追跡することで,タンパク質のリン酸化状態を推定する研究も行われています。しかし,タンパク質のリン酸化状態は同じタンパク質であっても一定であるとは限らず,同じ生理条件下においても複数のリン酸化フォームが生じていることがほとんどです。そのため,タンパク質そのものではなく,酵素消化した後のペプチドの分析を行う質量分析装置を用いた解析法,いわゆる「ボトムアッププロテオミクス法」では,タンパク質のリン酸化状態を正確に理解することは難しいとされます(図1)。
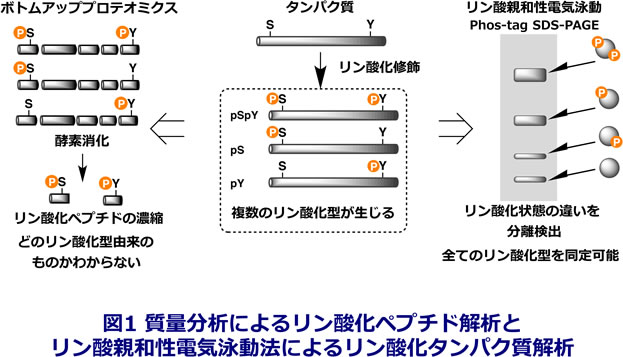
私たちはこれまでに,タンパク質のリン酸化状態を解析する手法として,リン酸基を特異的に捕捉するフォスタグ1)を用いたリン酸親和性電気泳動法を開発しています2-8)。この手法では,同じタンパク質であっても,リン酸化部位やリン酸基の数によってタンパク質の移動度が異なります(図1)。そのため,リン酸化状態の異なるタンパク質種を貴重な分子量情報を失うことなくネイティブサイズのまま分離検出でき,リン酸化フォームごとの時間的・空間的な変動をとらえることが可能となります。しかしながらこの手法は,ごく微量なリン酸化タンパク質が標的の場合,抗体を用いたウェスタン解析の併用が必須であり,未知のリン酸化部位の同定のためには,リン酸化タンパク質を網羅的に分取・濃縮する高精度,かつ,高効率なサンプル前処理技術が非常に重要となります。そこで本研究では,磁気ビーズを用いたマグネット技術とフォスタグ技術を融合させることで,迅速性と簡便性の点でこれまでにない高精度,かつ,高効率なアフィニティー濃縮法を創出します。
生体試料においてごく微量にしか存在しないリン酸化タンパク質を生体内の環境に近い条件で濃縮することは非常に有用な手法となります。そうした観点から,本研究では以下の2点を目標におき,生理的(中性)pHでリン酸基を捕捉するというフォスタグの特性を利用したタンパク質リン酸化研究のニーズに応えるべき高精度,かつ,高効率な分析技法を実現します。
- 与えられた磁場の内部で強力な磁力を発揮するマイクロサイズの磁気ビーズを基材として,それにフォスタグを固定化させた新規分子デバイスを開発します。
- 上記の新規分子デバイスを利用することで,細胞内において様々なリン酸化状態で混在するリン酸化タンパク質群を,それらの分子量情報を失わずに分取・濃縮するための簡便・高効率,かつ,高精度なアッセイ法を確立します。
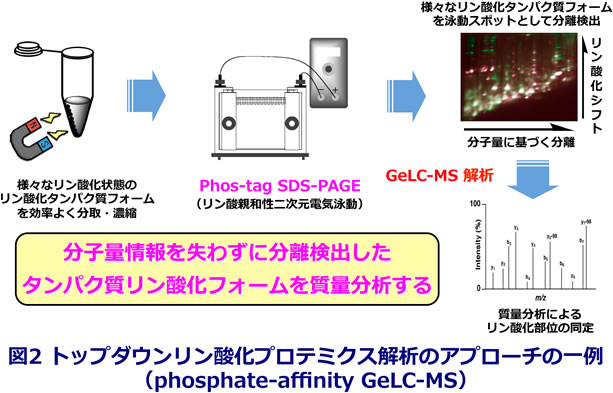
これらの目標を達成することで「トップダウンリン酸化プロテオミクス」を指向した解析アプローチを構築します(図2)。
【参考文献】
- Eiji Kinoshita, Takahashi, M., Takeda, H., Shiro, M., and Koike, T. Recognition of phosphate monoester dianion by an alkoxide-bridged dinuclear zinc(II) complex. Dalton Transaction, 1189–1193 (2004).
- Eiji Kinoshita, Kinoshita-Kikuta E., Takiyama, K., and Koike, T. Phosphate-binding tag, a new tool to visualize phosphorylated proteins. Molecular & Cellular Proteomics, 5:749–757 (2006).
- Eiji Kinoshita, Kinoshita-Kikuta, E., and Koike, T. Separation and detection of large phosphoproteins using Phos-tag SDS-PAGE. Nature Protocols, 9:1513–1521 (2009).
- Eiji Kinoshita, and Kinoshita-Kikuta E. Improved Phos-tag SDS-PAGE under neutral pH conditions for advanced protein phosphorylation profiling. Proteomics, 11: 319–323 (2011).
- Eiji Kinoshita, Kinoshita-Kikuta E., and Koike T. Phos-tag SDS-PAGE systems for phosphorylation profiling of proteins with a wide range of molecular masses under neutral pH conditions. Proteomics, 12:192–202 (2012).
- 木下英司 編 日本電気泳動学会誌 生物物理化学,56巻 Supplement 1
特集号「リン酸化生体分子解析のためのフォスタグテクノロジー」s1–s75 2012年 - 木下英司,木下恵美子,小池透,リン酸化プロテオミクスのための Phos-tag テクノロジー.
日本分析化学会誌 分析化学,61巻 特集号「オミクスと分析化学」,469–487 2012年 - 木下英司,木下恵美子,小池透,タンパク質リン酸化修飾の高感度検出法を新しいオミクス技術としての展開.
日本生化学会誌 生化学,86巻 特集号「次世代シグナル研究 —先駆的基礎解析と臨床・創薬への展開—」,447–455 2013年











