
CYLDによるK63結合型および直鎖型ポリユビキチン鎖選択的切断機構の詳細な解析
 |
研究代表者 佐藤裕介 東京大学・放射光連携研究機構・生命科学部門・構造生物学研究室 http://www.iam.u-tokyo.ac.jp/srro/SRROLifeSciDivJp2/Top.html |
【研究概要】
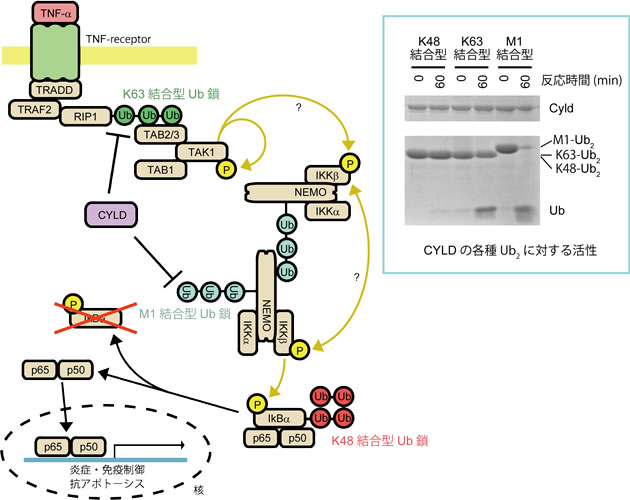
ユビキチン(Ub)が多数連なって形成されるポリUb鎖は様々な生体反応において重要なシグナルとして働き、その機能と構造についての知見は近年大きな進歩を遂げている。ポリUb鎖の形成に使われる残基は少なくとも8種類あり(Lys6, 11, 27, 29, 33, 48, 63及びMet1)、使われる残基によって異なるタンパク質機能制御様式をもつ。免疫・炎症反応の過程で刺激誘導される多くの遺伝子発現の発現誘導に関わるNF-κBシグナル伝達経路においては、Lys63結合型ポリUb鎖と、Met1結合型ポリUb鎖が活性化のシグナルとして重要な役割を果たすことが明らかとなっている(図)。
一方、家族性円柱腫症の原因遺伝子産物として同定された癌抑制タンパク質である脱Ub化酵素CYLDは、Lys63結合型ポリUb鎖とMet1結合型ポリUb鎖を特異的に切断することでNF-κB経路を負に制御し腫瘍形成を抑制する(図)。従ってCYLDの機能解明は腫瘍抑制メカニズムの理解につながるものであり、大きな注目を集めている。しかし、CYLDの機能発現において最も重要であると考えられるポリUb鎖の選択的切断メカニズムの詳細については明らかとされておらず解明が期待されている。そこで本研究では、CYLDとMet1結合型Ub鎖、およびK63結合型Ub鎖の複合体の構造を結晶構造解析の手法を用いて決定し、CYLDの癌抑制メカニズムの解明と共に、ポリUb鎖の識別メカニズムに対し構造学的なアプローチで詳細を明らかにする。
【参考文献】
- Bignell, G.R., Warren, W., Seal, S., Takahashi, M., Rapley, E., Barfoot, R., Green, H., Brown, C., Biggs, P.J., Lakhani, S.R., et al. (2000). Identification of the familial cylindromatosis tumour-suppressor gene. Nat Genet 25, 160-165.
- keda, F., and Dikic, I. (2006). CYLD in ubiquitin signaling and tumor pathogenesis. Cell 125, 643-645.
- Komander, D., Lord, C.J., Scheel, H., Swift, S., Hofmann, K., Ashworth, A., and Barford, D. (2008). The structure of the CYLD USP domain explains its specificity for Lys63-linked polyubiquitin and reveals a B Box module. Mol Cell 29, 451-464.
- Komander, D., Reyes-Turcu, F., Licchesi, J.D., Odenwaelder, P., Wilkinson, K.D., and Barford, D. (2009). Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Rep 10, 466-473.
- Kovalenko, A., Chable-Bessia, C., Cantarella, G., Israel, A., Wallach, D., and Courtois, G. (2003). The tumour suppressor CYLD negatively regulates NF-kappaB signalling by deubiquitination. Nature 424, 801-805.
- Sato, Y., Yoshikawa, A., Mimura, H., Yamashita, M., Yamagata, A., and Fukai, S. (2009a). Structural basis for specific recognition of Lys 63-linked polyubiquitin chains by tandem UIMs of RAP80. EMBO J.
- Sato, Y., Yoshikawa, A., Yamagata, A., Mimura, H., Yamashita, M., Ookata, K., Nureki, O., Iwai, K., Komada, M., and Fukai, S. (2008). Structural basis for specific cleavage of Lys 63-linked polyubiquitin chains. Nature 455, 358-362.
- Sato, Y., Yoshikawa, A., Yamashita, M., Yamagata, A., and Fukai, S. (2009b). Structural basis for specific recognition of Lys 63-linked polyubiquitin chains by NZF domains of TAB2 and TAB3. EMBO J 28, 3903-3909.
- Tokunaga, F., Sakata, S., Saeki, Y., Satomi, Y., Kirisako, T., Kamei, K., Nakagawa, T., Kato, M., Murata, S., Yamaoka, S., et al. (2009). Involvement of linear polyubiquitylation of NEMO in NF-kappaB activation. Nat Cell Biol 11, 123-132.
- Tokunaga, F., Nishimasu, H., Ishitani, R., Goto, E., Noguchi, T., Mio, K., Kamei, K., Ma, A., Iwai, K., and Nureki, O. (2012). Specific recognition of linear polyubiquitin by A20 zinc finger 7 is involved in NF-kappaB regulation. EMBO J. 31, 3856-3870.











