
シミュレーションを援用したシグナル伝達系による動的細胞形態制御の解析技術の開発
 |
研究代表者 行縄直人 京都大学 大学院情報学研究科 http://hawaii.sys.i.kyoto-u.ac.jp/ |
【研究概要】
分子シグナルによる細胞骨格の動的な制御は、生命の本質に関わる重要な仕組みである。細胞には多くの構成要素が含まれ、それらは相互作用しながら全体として協調してはたらくことで様々な環境に対して柔軟な応答を見せるが、その動作原理には未解明な点が多い。また、非常に動的な現象であるため、分子イメージング技術が目覚しい発展を見せている昨今においても、実験的に複数の分子の動態を同時かつ十分な時空間分解能で定量的に捉えることは難しい。したがって、そのシステム的理解のためには、計算機シミュレーションによる外挿(予測)と実験データの統計的解析による内挿(システム同定)を組み合わせたデータ同化技術が必要不可欠である。本研究課題では、低分子量GTP結合タンパク質Rhoファミリーに関するシグナル伝達ネットワークによる細胞形態制御機能に注目し、シグナル伝達におけるいかなる要因が細胞形態変化にいかに寄与するかについて、分子動態と形態情報の定量的理解を目指し、1)数理モデルの構築・シミュレーション技術、および、2)統計的データ解析の2つの方法でアプローチする。
1. 大規模シミュレーションによる細胞形態制御の包括的理解
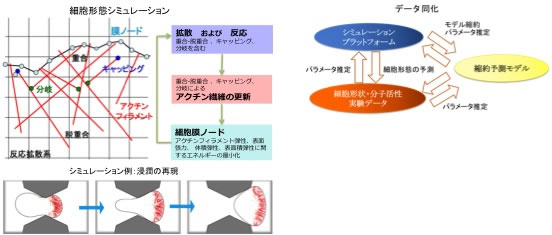
我々は、単一細胞に関する、1)RhoAを含むシグナル分子の反応拡散系、2)重合・脱重合・キャッピング・分枝を考慮した骨格系の確率的反応、3)骨格系(アクチンフィラメント)と膜との物理的相互作といった3つの細胞形態変化に関わる素過程に着目し、これらを統合したシミュレーションを行うためのプラットフォームの開発を進めてきた(図左)。本研究課題では、これをさらに拡張し、対象としているRhoファミリー低分子量Gタンパク質系シグナル伝達経路、および、リンカー蛋白質による架橋を含めたアクチンフィラメントのキネティクスの詳細化を行う。これにより、特徴的な局所細胞骨格構造形成のシミュレーションによる定性的な再現を試みる。
2. データ同化に基づく実験データからの細胞形態制御因子の同定
前述の通り、昨今の分子イメージングの技術を用いたとしても、細胞形態変化に関わると考えられる分子を網羅的かつ高い時空間分解能で捉えることは困難である。一方、シグナル伝達経路に含まれる分子を網羅したシミュレーションを用いることで、あくまでもモデルに含まれる分子に限られるが、定量的な情報を任意の分解能で得ることが可能である。この性質を利用し、実験で得られた不完全な観測値を補完するためことを考える。すなわち、実験による観測値と我々が開発したシミュレーションプラットフォームによる詳細な予測値とをデータ同化により統合することで、システムの包括的な理解を図る(図右)。また、シミュレーションで得られる「完全」な時空間データから、細胞形態変化に貢献する要因を統計的解析により絞り込むことで、一部の分子の動態から形態を予測する、縮約予測モデルの導出を試みる。
【参考文献】
- Nonaka,
S., Naoki, H., and Ishii, S.: A multiphysical model of cell migration
integrating reaction-diffusion, membrane and cytoskeleton. Neural Networks (to appear in 2011).
- Yukinawa, N., Yoshioka,
T., Kobayashi, K., Ogasawara, N., and Ishii, S.: A constrained Gaussian mixture
model for correlation-based cluster analysis of gene expression data. IPSJ Transactions on Bioinformatics,
vol. 2, 47-62 (2009).
- Yukinawa, N., Oba, S., Kato, K., and *Ishii, S.: Optimal aggregation of
binary classifiers for multi-class cancer diagnosis using gene expression
profiles. IEEE/ACM Transactions on
Computational Biology and Bioinformatics, vol. 6, no. 2, 333-343 (2009).
- Naoki, H., Sakumura, S., and Ishii, S.: Stochastic control of spontaneous
signal generation for gradient sensing in chemotaxis. Journal of Theoretical Biology, 255, 259-266 (2008).
- Yukinawa, N., Oba, S., *Kato, K., Taniguchi, K., Iwao-Koizumi, K., Tamaki,
Y., Noguchi, S., and Ishii S.: A multi-class predictor based on a probabilistic
model: application to gene expression profiling-based diagnosis of thyroid tumors. BMC Genomics, 7(1):190 (2006).
- Yukinawa, N., Yoshimoto, J., Oba, S., and Ishii, S.: System identification of gene expression time-series based on a linear dynamical system model with variational Bayesian estimation. IPSJ Transactions on Mathematical Modeling and Its Applications. 46(10), 57-65 (2005).











