
チューブリンチロシンリガーゼとチューブリン複合体の構造解析
 |
研究代表者 三島正規 首都大学東京 理工学研究科 http://www.comp.tmu.ac.jp/osbc2/ |
【研究概要】
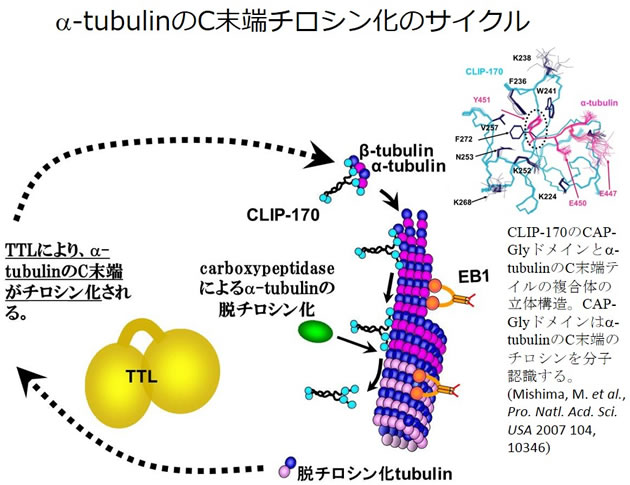
微小管は動的な性質を持ち、染色体分配や、細胞内輸送におけるレールとしての役割など、様々な場面で生命にとって重要な機能を担っています。近年、微小管の伸長端(プラス端)に集積し、その伸長を促進するplus-end tracking proteins(+TIPs)と総称される微小管結合タンパク質の研究が急速に発展しつつあり、我々は重要な+TIPsであるEB1の微小管結合部位の解析を行いました(Kanaba, T. et al., Biochim Biophys Acta. (2013)1834, 499-507 )。また同じく+TIPsであるCLIP-170が、α-チューブリンのC末端のチロシンを分子認識し、結合する際の構造基盤を明らかにしています(Mishima, M. et al., Pro. Natl. Acd. Sci. USA 2007 104, 10346)。
興味深いことに、このα-チューブリンのC末端チロシンは、脱チロシン化とチロシン化のサイクルを持ち、チロシン化の状態の違いが微小管動態や機能の制御にとって重要であることが指摘されています。またチロシン化のサイクルにおいて、特異的にα-チューブリンのC末端にチロシンを付加する酵素、Tubulin Tyrosine Ligase(TTL)が存在し、最近、この酵素のKOマウスが致死であること、神経細胞の機能に重要であることが明らかになり(Pro. Natl. Acd. Sci. USA 2005, 102, 7853)、その分子レベルでの理解が極めて重要な問題となってきています。またTTLは、Tubulin Tyrosine Ligase-Like protein(TTLL)と呼ばれる一連の酵素とファミリーを形成し、他のTTLLではtubulinへのポリグリシン付加、ポリグルタミン酸付加を行うものもあり、微小管の化学修飾による多様な制御機構の存在が明らかになりつつあります。したがって、TTLやTTLLの立体構造、あるいはこれらとチューブリンとの複合体の立体構造の知見は、微小管の化学修飾による制御機構を理解するために非常に大切です。我々はこれらの立体構造解析、相互作用解析に取り組みます。
また、他のシステムにおいても、リン酸化による制御などの修飾シグナルの構造研究について研究を進めています。これらについても本領域で活発に議論したいと思います。また共同研究まで発展するようなことがあれば幸いです。
参考文献
- “Structure of the second RRM domain of Nrd1, a fission yeast MAPK target RNA binding protein, and implication for its RNA recognition and regulation”
Kobayashi A, Kanaba T, Satoh R, Fujiwara, T, Ito Y, Sugiura R, Mishima M
Biochem. Biophys. Res. Commun. (2013) in press - “High-resolution heteronuclear multidimensional NMR of proteins in living insect cells using a baculovirus protein expression system.”
Hamatsu J, O'Donovan D, Tanaka T, Shirai T, Hourai Y, Mikawa T, Ikeya T, Mishima M, Boucher W, Smith BO, Laue ED, Shirakawa M, Ito Y
J. Am. Chem. Soc. 135, 1688-1691 (2013) - “Microtubule-binding sites of the CH domain of EB1 and its autoinhibition revealed by NMR”
Kanaba T, Maesaki R, Mori T, Ito Y, Hakoshima T, Mishima M.
Biochim Biophys Acta. 1834, 499-507 (2013) - “NMR assignments of SPOC domain of the human transcriptional corepressor SHARP in complex with a C-terminal SMRT peptide.”
Mikami S, Kanaba T, Ito Y, Mishima M.
Biomol NMR Assign. 2012 Sep 18. [Epub ahead of print] - “Insights into substrate recognition by the Escherichia coli Orf135 protein through its solution structure.”
Kawasaki K, Kanaba T, Yoneyama M, Murata-Kamiya N, Kojima C, Ito Y, Kamiya H, Mishima M.
Biochem Biophys Res Commun. 420, 263-268 (2012) - “Solution structure of the HRDC domain of human Bloom syndrome protein BLM.”
Sato A, Mishima M, Nagai A, Kim SY, Ito Y, Hakoshima T, Jee JG, Kitano K.
J Biochem. 148, 517-25 (2010) - “Protein structure determination in living cells by in-cell NMR spectroscopy”
Sakakibara D, Sasaki A, Ikeya T, Hamatsu J, Hanashima T, Mishima M, Yoshimasu M, Hayashi N, Mikawa T, Wälchli M, Smith BO, Shirakawa M, Güntert P, Ito Y.
Nature 458, 102-105 (2009) - “Structural basis for tubulin recognition by cytoplasmic linker protein 170 and its autoinhibition.”
Mishima M, Maesaki R, Kasa M, Watanabe T, Fukata M, Kaibuchi K, Hakoshima T.
Proc Natl Acad Sci U S A. 104, 10346-10351 (2007) - “Small-molecule ligand induces nucleotide flipping in (CAG)n trinucleotide repeats.”
Nakatani K, Hagihara S, Goto Y, Kobori A, Hagihara M, Hayashi G, Kyo M, Nomura M, Mishima M, Kojima C.
Nat Chem Biol. 1 39-43 (2005) - “Structure of human MTH1, a Nudix family hydrolase that selectively degrades oxidized purine nucleoside triphosphates.”
Mishima M, Sakai Y, Itoh N, Kamiya H, Furuichi M, Takahashi M, Yamagata Y, Iwai S, Nakabeppu Y, Shirakawa M.
J Biol Chem. 279, 33806-33815 (2004)











