
TRAF6複合体によるシグナル伝達の構造学的解明
 |
研究代表者 中村照也 熊本大学大学院生命科学研究部 http://www.pharm.kumamoto-u.ac.jp/Labs/sb/ |
【研究概要】
近年,細胞内シグナル伝達において,従来のリン酸化のみならずユビキチン化などの新たな修飾が関与するという報告例が増加しており,シグナル伝達の厳密かつ複雑な分子基盤が明らかにされつつある.免疫応答やアポトーシスなどの遺伝子制御に関わる転写因子NF-κBの活性化経路では,ユビキチン化を介したシグナル伝達の主要因子の1つとしてTRAF6が重要な役割を担っている.
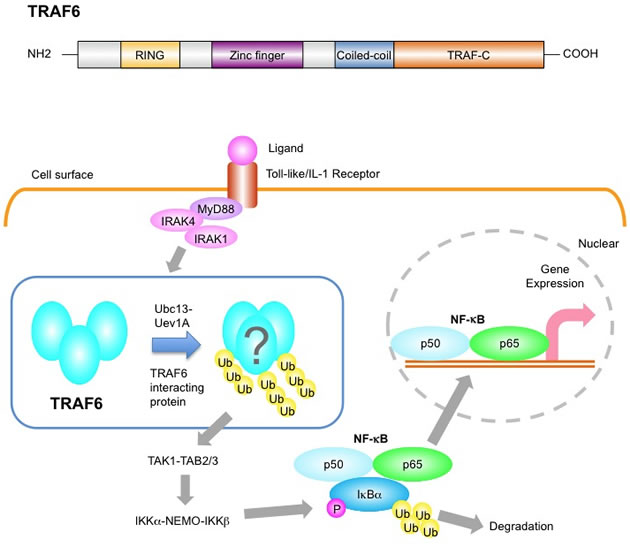
本課題では,NF-κBの活性化に関与するTNFとTNFR2複合体の構造解析に続き,そのTNFR系のみならずTLR/IL-1R系にも関わるというユニークな特徴を持つTRAF6のシグナル伝達に注目した.TRAF6は,N末端から①自己ポリユビキチン化に関わるRING・Zincフィンガードメイン,②自身の3量体化に関わるCoiled-coil領域,③他の分子との相互作用に必要なTRAF-Cドメインの3つの領域から構成される (図).TRAF6は,TLR/IL-1Rからの刺激を受けて,自身のユビキチンリガーゼ活性 (E3活性) により自己ポリユビキチン化を行い,IKK活性化によるIκBのリン酸化と分解を経て,最終的にNF-κBを活性化すると考えられている (図).このTRAF6の自己ポリユビキチン化は,他のアダプター分子との相互作用を介してTRAF6の会合状態が変化することで引き起こされると考えられている.TRAF6の構造学的研究では,Coiled-coil領域を含まないTRAF-Cドメインの構造およびRING・ZincフィンガードメインとUbc13 (ユビキチン結合酵素) との複合体構造などの部分構造のみが明らかにされており,TRAF6が他の分子とどのような結合を基盤として,NF-κBの活性化に重要な自己ポリユビキチン化を誘起するのかという構造学的知見は未だ不明である.本研究では,TLR/IL-1Rを介するNF-κB活性化シグナル経路におけるTRAF6複合体およびTRAF6との結合に関わるアダプター分子のX線結晶構造解析を行い,TRAF6が行う自己ポリユビキチン化を介したシグナル伝達の構造学的基盤を解明する.
参考文献
- Nakamura, T., Zhao, Y., Yamagata, Y., Hua, Y. J., Yang, W. Mechanism of the nucleotidyl-transfer reaction in DNA polymerase revealed by time-resolved protein crystallography. Biophysics, 9, 31-36 (2013)
- Koga, Y., Inazato, M., Nakamura, T., Hashikawa, C., Chirifu, M., Michi, A., Yamashita, T., Toma, S., Kuniyasu, A., Ikemizu, S., Nakabeppu, Y., Yamagata, Y. Crystallization and preliminary X-ray analysis of human MTH1 with a homogeneous N-terminus. Acta Cryst. F69, 45-48 (2013)
- Nakamura, T., Zhao, Y., Yamagata, Y., Hua, Y. J., Yang, W. Watching DNA polymerase η make a phosphodiester bond. Nature, 487, 196-201 (2012)
- Arimori, T., Tamaoki, H., Nakamura, T., Kamiya, H., Ikemizu, S, Takagi, Y, Ishibashi, T, Harashima, H, Sekiguchi, M, Yamagata, Y. Diverse substrate recognition and hydrolysis mechanisms of human NUDT5. Nucleic Acids Res., 39, 8972-8983 (2011)
- Nishi, K., Ono, T., Nakamura, T., Fukunaga, N., Izumi, M., Watanabe, H., Suenaga, A., Maruyama, T., Yamagata, Y., Curry, S., Otagiri, M. Structural insights into differences in drug binding selectivity between two forms of human alfa1-acid glycoprotein genetic variants, the A and F1*S forms. J. Biol. Chem., 286, 14427-34 (2011)
- Mukai, Y., Nakamura, T., Yoshikawa, M., Yoshioka, Y., Tsunoda, S., Nakagawa, S., Yamagata, Y., Tsutsumi, Y. Solution of the Structure of the TNF-TNFR2 Complex. Science Signaling, 3, ra83 (2010)
- Nakamura, T., Meshitsuka, S., Kitagawa, S., Abe, N., Yamada, J., Ishino, T., Nakano, H., Tsuzuki, T., Doi, T., Kobayashi, Y., Fujii, S., Sekiguchi, M., Yamagata, Y. Structural and dynamic features of the MutT protein in the recognition of nucleotides with the mutagenic 8-oxoguanine base. J. Biol. Chem., 285, 444-452 (2010)
- Mukai, Y., Nakamura, T., Yoshioka, Y., Shibata, H., Abe, Y., Nomura, T., Taniai, M., Ohta, T., Nakagawa, S., Tsunoda, S., Kamada, H., Yamagata, Y., Tsutsumi, Y. Fast binding kinetics and conserved 3D structure underlie the antagonistic activity of mutant TNF: useful information for designing artificial proteo-antagonists. J. Biochemistry, 146, 167-172 (2009)
- Mukai, Y., Nakamura, T., Yoshioka, Y., Tsunoda, S., Kamada, H., Nakagawa, S., Yamagata, Y., Tsutsumi, Y. Crystallization and preliminary X-ray analysis of the tumour necrosis factor alpha-tumour necrosis factor receptor type 2 complex. Acta Cryst. F65, 295-298 (2009)
- Mukai, Y., Shibata, H., Nakamura, T., Yoshioka, Y., Abe, Y., Nomura, T., Taniai, M., Ohta, T., Ikemizu, S., Nakagawa, S., Tsunoda, S., Kamada, H., Yamagata, Y., Tsutsumi, Y. Structure-function relationship of tumor necrosis factor (TNF) and its receptor interaction based on 3D structural analysis of a fully active TNFR1-selective TNF mutant. J. Mol. Biol., 385, 1221-1229 (2009)











